摘要:抗菌肽(AMP)作为未来最有希望解决病原微生物耐药性的新型抗菌药物之一,其研发备受关注。抗菌肽一般较短,组成多样,迄今人们已发现数千条天然抗菌肽,并建立了多个公开的抗菌肽数据库,为新型抗菌肽的研发和设计奠定了基础。在抗菌肽的信息描述方面,人们使用了伪氨基酸残基组成,、位置特异性评分矩阵、独热编码等多种特征向量。在深度学习方法上,研究人员应用了RNN、CNN、GNN等多种算法,开发了ACEP、CLaSS等抗菌肽活性预测和序列生成模型。这些模型有望加速新型抗菌肽的发现,为应对耐药菌感染,尤其是临床上难以治疗的耐药性革兰阴性菌感染,提供新的手段。
一份抗生素耐药性的评估报告指出,2050年可能有1000万人死于耐药细菌感染,虽然目前上市的抗生素对治疗绝大多数感染依然有效,但由于人类长期使用抗生素,越来越多的耐药菌出现,还有很多多重耐药菌造成了医院许多严重感染。目前作为治疗耐药菌的最后手段的碳青霉烯类抗生素也开始面临耐药性问题,因此,需要新的抗菌药物来应对这一问题
抗菌肽(antimicrobial peptide, AMP)是最有希望 解决耐药菌问题的新型抗菌药物之一,序列一般较 短,组成变化多样,但多为阳离子两亲性多肽分 子,其抗菌作用具有多种可能机制,其中最常见的 是通过与带负电荷的脂多糖(革兰阴性)或脂磷壁酸 (革兰阳性)的磷酸基团的静电相互作用到达细胞膜, 以库仑力吸附于细胞膜或进入细胞,随后膜破裂、 细胞质渗漏,导致细菌死亡。抗菌肽通过靶向整 个细胞成分,而不是特定的分子,具有广谱的抗菌 活性,同时避开了碳青霉烯类和替加环素等单一 靶点药物的耐药性机制,该生化特性和药效学性质 使其比传统抗生素更难耐药
可惜的是,虽然迄今人们已发现成千上万条天然抗菌肽,且已有多个 抗菌肽数据库被建立并公开,但是一方面由于抗生 素新药研发耗时、昂贵、失败率高且盈利空间小, 新抗菌药物的研发进入了冷门期,大型制药公司已 基本放弃该市场,另一方面因抗菌肽结构不稳定 性、多肽易降解和非特异性膜裂解的体内毒性等因 素,限制了抗菌肽临床使用。
Stokes等 就利用深度神经网络从1.07亿个分子发现了在小鼠体 内有广谱抗菌活性的新抗生素halicin,使用这一方法 可在4 d内完成十多亿化合物分子的虚拟筛选,其效 率远超传统的筛选手段。这是一种可以低成本、高 效地发现活性高、毒性低以及结构稳定能临床应用 的抗菌肽的新方法。
1.收集数据集

上图展现了深度学习处理抗菌肽数据的一般过程,前两步中数据来源与数据处理的方法明显区别于深度学习在其他方面的应用,且不同的神经网络模型具有不同的用途
-
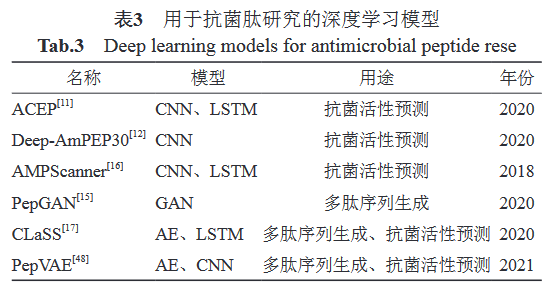
GAN和AE常被用于抗菌肽序列生成,有事RNN也会被用于序列生成
Tucs A, Tran D P, Yumoto A, et al. Generating ampicillinlevel antimicrobial peptides with activity-aware generative adversarial networks[J]. ACS Omega, 2020, 5(36): 22847-22851.
Dean S N, Walper S A. Variational autoencoder for generation of antimicrobial peptides[J]. ACS Omega, 2020, 5(33): 20746-20754.
Das P, Sercu T, Wadhawan K, et al. Accelerated antimicrobial discovery via deep generative models and molecular dynamics simulations[J]. Nat Biomed Eng, 2021, 5(6): 613-623.
Wang C, Garlick S, Zloh M. Deep learning for novel antimicrobial peptide design[J]. Biomolecules, 2021, 11(3): 471.
-
CNN和RNN常被用于抗菌肽的活性预测。
Fu H, Cao Z, Li M, et al. ACEP: Improving antimicrobial peptides recognition through automatic feature fusion and amino acid embedding[J]. BMC Genomics, 2020, 21(1): 597.
Yan J, Bhadra P, Li A, et al. Deep-AmPEP30: Improve short antimicrobial peptides prediction with deep learning[J]. Mol Ther-Nucl Acids, 2020, 20: 882-894.
Daniel V, Uday K, Amarda S. Deep learning improves antimicrobial peptide recognition[J]. Bioinformatics, 2018, 34(16): 2740-2747.
Müller A T, Hiss J A, Schneider G. Recurrent neural network model for constructive peptide design[J]. J Chem Inf Model, 2018, 58(2): 472-479.
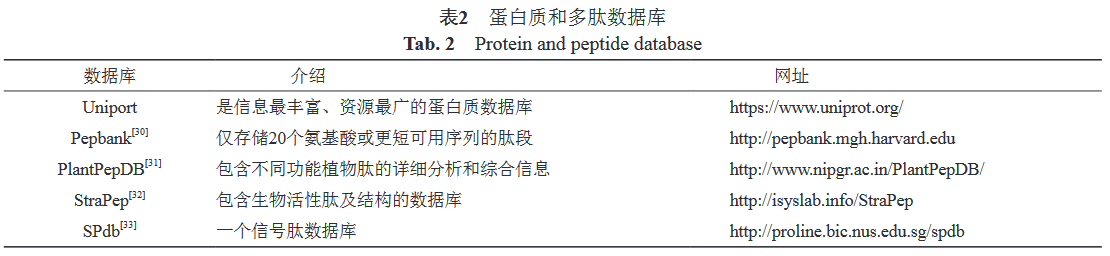
下图展示一些通用抗菌肽数据库及其相关信息,更多特定的抗菌肽数据库可以查看文献。

有监督学习:抗菌肽活性预测的二分类问题,标签通常是诱惑性或无活性,阳性数据集常在抗菌肽数据库中获得,阴性在其他蛋白质多肽数据库中获得。
无监督学习:Das等就设计 了可以在UniProt数据库中报告的所有肽序列(可能无 注释)上训练的无监督学习模型。
Das P, Sercu T, Wadhawan K, et al. Accelerated antimicrobial discovery via deep generative models and molecular dynamics simulations[J]. Nat Biomed Eng, 2021, 5(6): 613-623.
2.数据预处理
抗菌肽特征 构建不仅借鉴计算机科学处理序列问题时使用的独 热编码(one-hot encoder)、特征张量嵌入(feature tensor embedding)和Word2vec词嵌入等方式,同时还伴随 生物信息学和计算生物学的进步产生更复杂和更具 描述性的特征,这些特征不仅与理化性质有关,而 且与微观层面的顺序结构以及进化信息等有关,如
-
氨基酸组成(amino acid composition, AAC),
-
伪氨基 酸组成(pseudo amino acid composition, PseAAC),
-
位置特异性评分矩阵(position-specific scoring matrix, PSSM)等。
独热编码作为一种多肽序列特征能在一定程度上反应多肽的序列信息,但它数据过于离散,很难捕捉到氨基酸之间的相似之处和不同之处。
特征张量嵌入则能够很好的解决这一点,他利用概率生成张量对氨基酸残基编码,该编码称为模型可训练的一部分,将氨基酸映射到可训练的实数张量,使用反向 传播算法不断更新这些实数张量,氨基酸之间的相似 性和差异性便可通过张量之间的几何距离来度量
Word2vec词嵌入是自然语言处理中的一种网络模 型,基于从大量文档语料库中收集邻近的单词数 据,通过训练数据所学得的参数,即隐层的权重矩 阵,生成该词语具有上下文属性的嵌入特征向量, 其中类似向量往往分配给出现在类似上下文中的单 词。Hamid等把多肽序列中的连续3个氨基酸作为 一个“词”,然后利用Word2vec中的skip-gram模型 生成的词嵌入向量,用于细菌素识别
氨基酸组成是Nakashima和Nishikawa在1994年 提出的,它现在一般指多肽序列中20种氨基酸分别 出现的频率,是一个有20个组分的向量
在此基 础上,发展出了伪氨基酸组成,其利用位置间隔为λ 的氨基酸的疏水性值、亲水性值以及侧链质量等(都 进行归一化处理)计算λ阶相关系数(θλ),若以向量X 表示多肽的伪氨基酸组成,那么X中的前20个组分是 归一化处理后第i种氨基酸出现频率ƒi(i=20),反映了 氨基酸组成的影响,后λ个元素是归一化处理后有一 定权重值ω的θλ,反映了氨基酸顺序和理化性质的 影响。
现人们可以通过网页服务器http://chou.med. harvard.edu/bioinf/PseAA/生成所需的PseAAC。在 PseAAC的基础上,还发展出了伪K-tuple减少氨基酸 组成(pseudo K-tuple reduced amino acids composition, PseKRAAC)等方法
位置特异性评分矩阵(PSSM)是进化信息的一种 常见表示[42],一个长为L的多肽序列中,其每一个 位置氨基酸突变为20种氨基酸的概率就构成了大小 为L×20的PSSM矩阵[43]。PSSM矩阵可以通过PSIBLAST程序获得,被Fu等[11]用于抗菌肽识别并获得 了不错的结果。
3.深度学习模型及应用
卷积神经网络CNN
可以有效地降 低网络的复杂度,减少训练参数的数目,使模型对 平移、扭曲、缩放具有一定程度的不变性,并具有 强鲁棒性和容错能力,且也易于训练和优化。Yan 等利用PseKRAAC和卷积神经网络开发了一个基 于序列的短AMP分类模型,称为Deep-AmPEP30, 该模型准确率比现有的基于机器学习的方法提高了 77%,并且发现了与氨苄青霉素活性相当的抗菌肽 P3(FWELWKFLKSLWSIFPRRRP)。
Yan J, Bhadra P, Li A, et al. Deep-AmPEP30: Improve short antimicrobial peptides prediction with deep learning[J]. Mol Ther-Nucl Acids, 2020, 20: 882-894.
循环神经网络RNN
Wang等搭建基于LSTM和双向LSTM的 模型成功地生成并筛选到可能具有抗大肠埃希菌活 性的新型AMPs
深度学习用于抗菌肽研究时,往往不局限于用 单一的神经网络分析抗菌肽数据集。Daniel等就 构建了一个包含嵌入层(embedding layer),卷积层 (convolutional layer),最大池化层(max pooling layer) 和LSTM层的深层神经网络模型,可以正确识别超过 98% APD 3数据库中的对革兰阳性或革兰阴性细菌具 有活性的AMP。
自动编码器AE
可通过编码器和解码器学习 输入分子特征(及其属性),然后在潜在数据空间进 行双向映射来生成新的分子,已被用于设计一个完 全自动化的计算框架CLaSS。CLaSS使用自动编码 器在多肽分子信息构建的潜在数据空间上进行训练,再利用线性插值的方法在空间中采样生成新的 多肽序列,然后使用深度学习分类器以及从高通量 分子动力学模拟得出的物理化学特征,来筛选生成 的多肽分子,可用于广谱的AMP序列的从头设计与 筛选。
生成对抗网络GNN
Tucs等 [15]设计的PepGAN 模型可以控制生成序列的概率分布,使之尽可能 多地覆盖活性肽,用该模型生成了一个最低抑 菌浓度仅为氨苄西林一半的高活性抗菌肽AMP4 (GLKKLFSKIKIGSALKNLA)
4.模型评估和不足
在计算上常使用灵敏度 (sensitivity)、特异性(specificity)、准确率(accuracy) 以及马修相关系数(matthews correlation coefficient, MCC)等作为评估指标,使用测试数据集来判断模型 的准确性。
但由于现有的抗菌肽特征表示方法,尚 难以完整地描述抗菌肽特征,也缺乏可以模拟和描 述AMP各种结构及物理化学特性的坚实理论[49],其 生成和预测结果并不完全可信,因此常常需要与其 他方法结合加以验证,比如Puentes等[50]提出了4种新 兴技术相结合的抗菌肽设计筛选流程,包括人工智 能、分子动力学、微生物表面展示(surface-display in microorganisms)和微流控(microfluidics),前两个是筛 选和设计的计算机策略,而后两个对应于实验方法 的合成和测试。使用实验合成并测试设计筛选的新 型抗菌肽的活性,可以更准确地评估模型的效果,同 时也可以发现一些有潜力的新抗菌肽。
5.总结与展望
上述多种特征构建方法可用于深度学习,以预 测抗菌活性或产生新的抗菌肽,但对于含复杂修饰 且未知空间结构的抗菌肽,如订书肽(即在多肽结构 中加入一个碳氢侧链或其他类型侧链以稳定其二级 结构的多肽)[52],尚缺乏合适的结构表征方法,并且 由于相应非天然抗菌肽的数据量较少,难以构建深 度学习模型
可喜的是,深度迁移学习以及图神经 网络等新的算法出现,有希望解决这些难题,
-
前者 可以在小数据集数据不足的情况下,先在大数据集上 预训练,然后在特定目标数据集(即小数据集)上微调 模型参数以实现模型在小数据集上的良好表现
-
后 者则是能将多肽分子中原子和键转变为节点与边的 图结构进行学习,实现对多肽复杂结构的表征,已 被用于多肽毒性的预测
-