一、选题背景
心血管疾病是全球范围内主要健康威胁之一,而通过大数据分析揭示相关模式和趋势对于疾病预防和管理至关重要。社会上心血管疾病的不断增加与现代生活方式、不良饮食和缺乏运动等因素密切相关,对健康系统和经济造成巨大负担。在技术飞速发展的今天,大数据分析为深入挖掘潜在的健康信息提供了前所未有的机会,借助医疗记录、生活方式数据和身体健康信息等多来源数据,能够更全面地了解心血管疾病的发病机制和影响因素。
二、大数据分析设计方案
1.数据集来源:
- 本次课程设计采用的数据集:心血管疾病数据集
- 数据集来源:爱数科,http://idatascience.cn/dataset-detail?table_id=29
2.数据集的数据内容:
| 字段名称 | 字段类型 | 字段说明 |
|---|---|---|
| id | 整型 | ID。 |
| age | 整型 | 年龄。 |
| gender | 整型 | 性别。 |
| height | 整型 | 身高。 |
| weight | 浮点型 | 体重。 |
| ap_hi | 整型 | 收缩压。 |
| ap_lo | 整型 | 舒张压。 |
| cholesterol | 整型 | 胆固醇,1:正常; 2:高于正常; 3:远高于正常。 |
| gluc | 整型 | 葡萄糖,1:正常; 2:高于正常; 3:远高于正常。 |
| smoke | 整型 | 病人是否吸烟。 |
| alco | 整型 | 酒精摄入量。 |
| active | 整型 | 体育活动。 |
| cardio | 整型 | 有无心血管疾病。 |
3.实现思路:
- 导入数据集;
- 数据清洗:缺失值、离群值处理,数据值转换计算;
- 统计数据集特征;
- 数据可视化分析;
- 建立预测模型。
4.技术难点:
- 数据清洗:统计数据特征,对离群值进行剔除;
- 数据可视化分析:实现回归图、核密度图、小提琴图、盒图等图,利用数据图进行数据可视化和信息分析;
- 构建决策树、随机森林的预测模型。
三、数据分析步骤
1、数据清洗
导入数据集,输出数据集维度和前5行数据:
1 import pandas as pd 2 3 CVD_data = pd.read_csv('F:\JupyterNotebookFile\data\心血管疾病数据集.csv') 4 print(CVD_data.shape) # 输出数据集的维度 5 CVD_data.head()
结果:
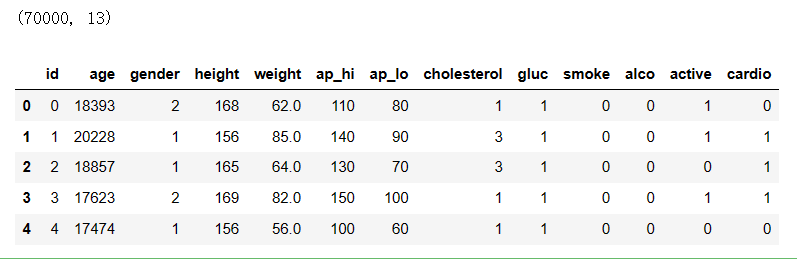
删除无用数据:
# id列的数据序列存在间断不连续,无法作为索引,因此无实际作用,可删除 delete_column = 'id' CVD_data.drop(delete_column, axis=1, inplace=True) print(CVD_data.shape) CVD_data.head()
结果:
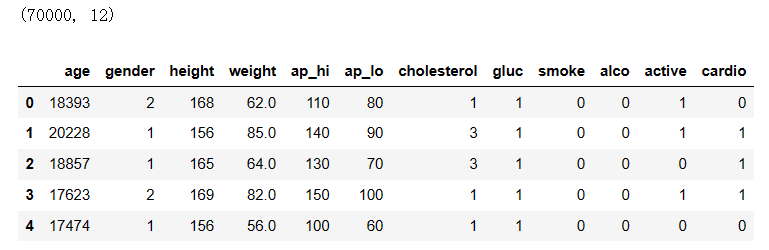
查找缺失值:
1 # 数据集信息 2 CVD_data.info()
通过输出数据集信息可以看出数据集没有缺失值,所以不需要进行缺失值处理:

由于年龄单位是天数,转为“年”更直观:
1 # 考虑到存在闰年和平年,取平均天数365.25天更精准,并且结果向下取整 2 import numpy as np 3 4 CVD_data['age'] = (np.floor(CVD_data['age'] / 365.25).astype(int)) 5 CVD_data['age'].head()
结果:

由于身高体重和心血管疾病也有很大关系,因此直接计算BMI值进行数据对比更为方便,计算后添加到数据集中:
1 # 计算BMI值,添加"BMI"列 2 CVD_data["BMI"] = (CVD_data["weight"] / (CVD_data["height"] / 100)**2).round(1) 3 CVD_data.sample(3)
结果:

将处理后的数据集保存为新文件,方便使用:
1 # 保存处理后的数据为新的CSV文件 2 CVD_data.to_csv('F:\JupyterNotebookFile\data\心血管疾病数据集(数据已处理).csv', index=False)
导入处理后的数据集并查看:
# 导入已处理完的数据集 CVD_data = pd.read_csv('F:\JupyterNotebookFile\data\心血管疾病数据集(数据已处理).csv') print(CVD_data.shape) # 输出数据集的维度 CVD_data.head()
结果:

统计数据的各个特征信息,包括样本数量、均值、最大值、最小值、四分位数:
# 对数据的各个特征进行统计 data_summary = CVD_data.describe() display(data_summary)
结果:

2、数据可视化分析
1 # 各个字段的数据分布情况 2 import matplotlib.pyplot as plt 3 CVD_data.hist(bins = 20, figsize = (40, 30)) 4 plt.show()
结果:
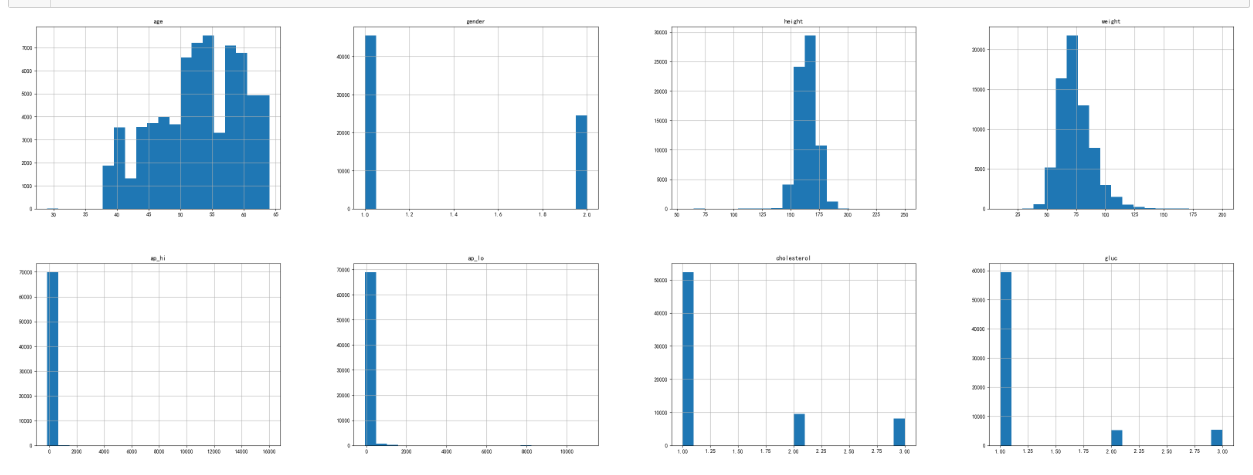
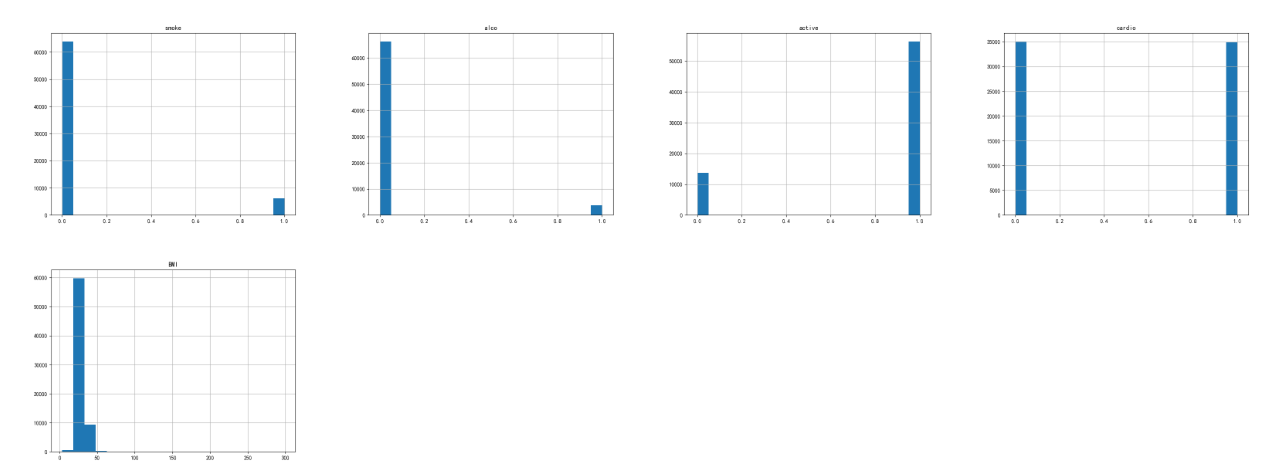
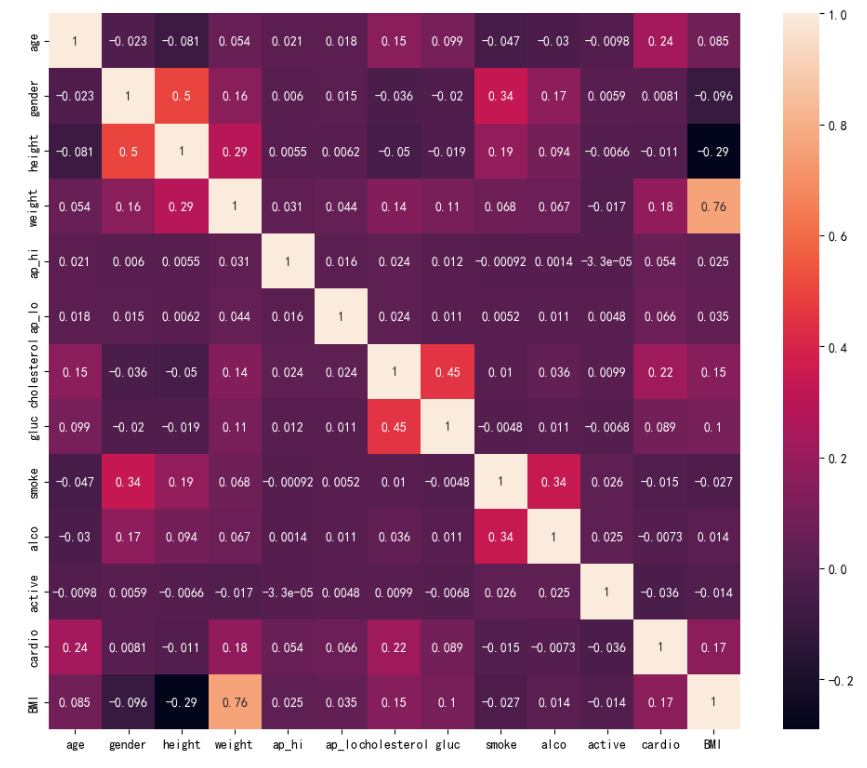
热力图显示各字段的相关性:
1 # 各字段的相关性 2 plt.figure(figsize = (12,10)) 3 sns.heatmap(CVD_data.corr(), annot =True) 4 plt.show()
结果:
1、患病、未患病,男性、女性占比分析:
1 # 患病、未患病,男性、女性占比分析 2 countHavaCVD = len(CVD_data[CVD_data.cardio == 1]) 3 countNoCVD = len(CVD_data[CVD_data.cardio == 0]) 4 countMale = len(CVD_data[CVD_data.gender == 1]) 5 countFemale = len(CVD_data[CVD_data.gender == 2]) 6 7 proportion_CVD = ("{:.2f}%".format((countHavaCVD / (len(CVD_data.cardio))*100))) 8 proportion_NoCVD = ("{:.2f}%".format((countNoCVD / (len(CVD_data.cardio))*100))) 9 proprotion_Male = ("{:.2f}%".format((countMale / (len(CVD_data.gender))*100))) 10 proportion_Female = ("{:.2f}%".format((countFemale / (len(CVD_data.gender))*100))) 11 12 # 创建包含计算结果的新 DataFrame 13 result_data = pd.DataFrame({ 14 ' ': ['样本数量', '占比'], 15 '患病': [countHavaCVD,proportion_CVD], 16 '未患病': [countNoCVD, proportion_NoCVD], 17 '男性': [countMale, proprotion_Male], 18 '女性': [countFemale, proportion_Female] 19 }) 20 21 display(result_data)
结果:

由数据可看出,患病人数占整个数据集样本数量的49.97%,数据集较为平衡,而本数据集中,有五分之三为男性样本,占到65.04%。
通过饼图可以更加直观地看出各自的样本数量占比:
1 import seaborn as sns 2 3 plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文标签 4 plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号 5 6 # 创建画布 7 fig = plt.figure(figsize = (8, 8)) 8 a = [countHavaCVD, countNoCVD] 9 b = [countMale, countFemale] 10 label1 = ['患病人数','未患病人数'] 11 label2 = ['男性','女性'] 12 #间隔 13 explode=(0,0.1) 14 #颜色 15 colors = ['#6a89cc','#1e3799'] 16 plt.subplot(1, 2, 1) 17 plt.pie(a,labels=label1, explode=explode ,colors=colors, autopct='%.2f%%',shadow=False,startangle=170) 18 plt.title('患病、未患病的人数比例') 19 #颜色 20 colors = ['#38ada9', '#78e08f'] 21 plt.subplot(1, 2, 2) 22 plt.pie(b,labels=label2,explode=explode ,colors=colors, autopct='%.2f%%',shadow=False,startangle=230) 23 plt.title('男性、女性的人数比例') 24 plt.show()
结果:

2、性别与患病的关系饼图:
# 计算男性患者在男性总人数中的占比 ratioMaleCVD = len(CVD_data[(CVD_data.gender == 1) & (CVD_data.cardio == 1)]) / countMale # 计算女性患者在女性总人数中的占比 ratioFemaleCVD = len(CVD_data[(CVD_data.gender == 2) & (CVD_data.cardio == 1)]) / countFemale fig = plt.figure(figsize = (8, 8)) labels_Male_CVD = ['男性患病', '男性未患病'] sizes_Male_CVD = [ratioMaleCVD, 1 - ratioMaleCVD] plt.subplot(1, 2, 1) plt.pie(sizes_Male_CVD, labels=labels_Male_CVD, colors=['#f1c400', '#f39c12'], autopct='%1.2f%%', startangle=50, explode=explode) plt.title('男性患病人数占比') labels_Female_CVD = ['女性患病', '女性未患病'] sizes_Female_CVD = [ratioFemaleCVD, 1 - ratioFemaleCVD] plt.subplot(1, 2, 2) plt.pie(sizes_Female_CVD, labels=labels_Female_CVD, colors=['#3F51B5', '#2196F3'], autopct='%1.2f%%', startangle=700, explode=explode) plt.title('女性患病人数占比') plt.show()
结果:

由饼图可以看出,男性和女性的患病几率基本相当,性别与患病几率没有多大关系。
3、年龄与患病情况的关系
1 # 统计每个年龄的未患病和患病人数 2 counts_noCVD = CVD_data[CVD_data['cardio'] == 0].groupby('age').size() 3 counts_CVD = CVD_data[CVD_data['cardio'] == 1].groupby('age').size() 4 5 plt.figure(figsize=(12, 14)) 6 # 绘制折线图 7 plt.subplot(3, 2, 1) 8 plt.plot(counts_noCVD.index, counts_noCVD.values, label='未患病', color='#ff793f') 9 plt.plot(counts_CVD.index, counts_CVD.values, label='患病', color='#2c2c54') 10 plt.title('年龄与患病人数关系') 11 plt.xlabel('年龄') 12 plt.ylabel('患病人数') 13 plt.legend() 14 # 绘制直方图 15 plt.subplot(3, 2, 2) 16 sns.histplot(x=CVD_data['age'], kde=True, color='#2c2c54') 17 plt.title('年龄与患病关系的直方图') 18 plt.xlabel('年龄') 19 plt.ylabel('人数') 20 # 绘制小提琴图 21 plt.subplot(3, 2, 3) 22 sns.violinplot(x='cardio', y='age', data=CVD_data, palette=['#ff793f', '#2c2c54']) 23 plt.title('年龄与患病关系的小提琴图') 24 plt.xlabel('患病情况') 25 plt.ylabel('年龄') 26 plt.subplot(3, 2, 4) 27 # 绘制核密度图 28 sns.kdeplot(x='age', data=CVD_data[CVD_data['cardio'] == 0], color='#ff793f', label='未患病', fill=True) 29 sns.kdeplot(x='age', data=CVD_data[CVD_data['cardio'] == 1], color='#2c2c54', label='患病', fill=True) 30 plt.title('年龄与患病关系的核密度图') 31 plt.xlabel('年龄') 32 plt.ylabel('核密度估计') 33 # 绘制盒图 34 plt.subplot(3, 2, 5) 35 sns.boxplot(x='cardio', y='age', data=CVD_data, palette=['#ff793f', '#2c2c54']) 36 plt.title('年龄与患病关系的盒图') 37 plt.xlabel('患病情况') 38 plt.ylabel('年龄') 39 # 绘制散点图 40 plt.subplot(3, 2, 6) 41 sns.regplot(x='age',y='cardio',data=CVD_data, color='#2c2c54') 42 plt.title('年龄与患病关系的散点图') 43 plt.xlabel('年龄') 44 plt.ylabel('患病情况') 45 plt.tight_layout() 46 plt.show()
结果
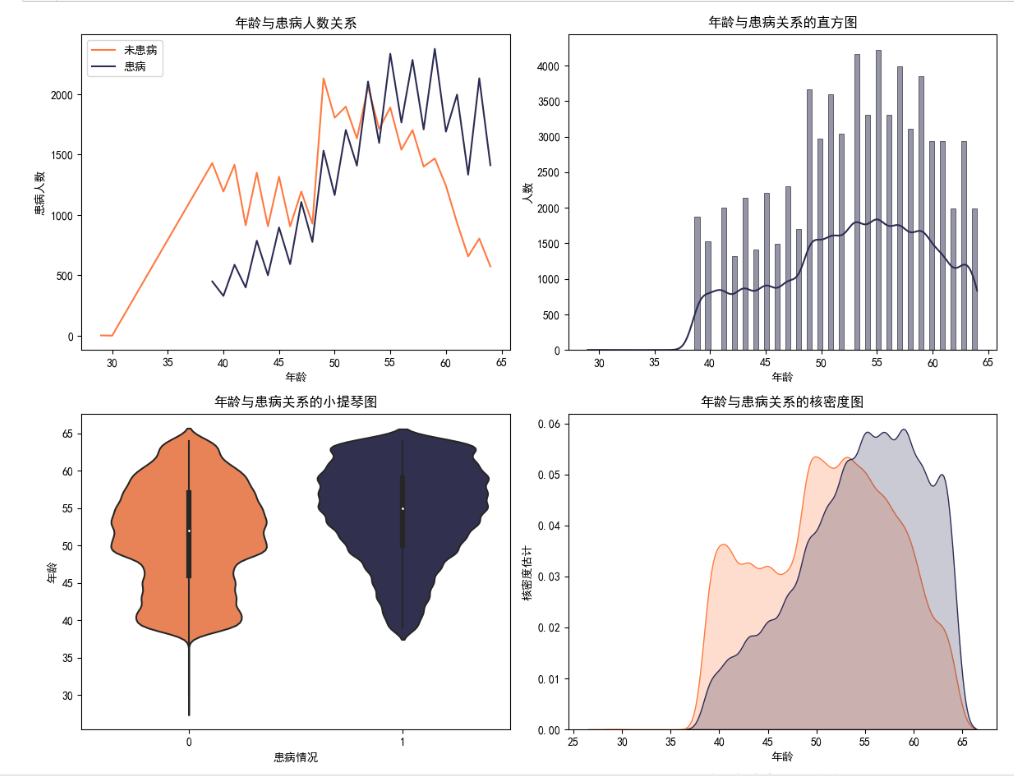
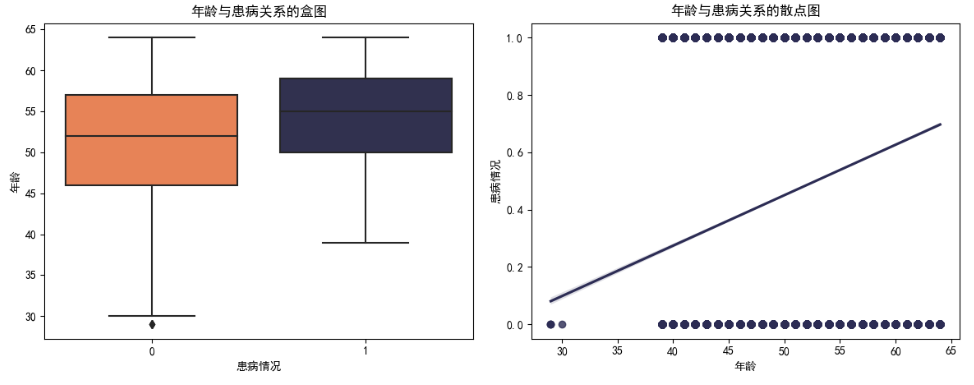
由以上图像可以看出,年龄越大,患病的概率越大,通过回归图可以更加清楚地看出趋势。核密度图也可以清楚地看出,从38岁开始,患病人数逐渐上升,在55岁达到峰值,而55岁到60岁之间有一段起伏,随后开始下降。
4.胆固醇、葡萄糖与患病情况的关系
1 plt.figure(figsize=(8, 6)) 2 # 绘制胆固醇——患病关系柱状图 3 plt.subplot(2, 2, 1) 4 sns.countplot(x=CVD_data['cholesterol'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 5 plt.title('胆固醇含量与患病关系') 6 plt.xlabel('胆固醇含量') 7 plt.ylabel('人数') 8 # 绘制胆固醇——患病关系回归图 9 plt.subplot(2, 2, 3) 10 sns.regplot(x='cholesterol', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 11 plt.title('回归图 - 胆固醇含量与患病关系') 12 plt.xlabel('胆固醇含量') 13 plt.ylabel('患病情况') 14 # 绘制葡萄糖——患病关系柱状图 15 plt.subplot(2, 2, 2) 16 sns.countplot(x=CVD_data['gluc'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 17 plt.title('葡萄糖含量与患病关系') 18 plt.xlabel('葡萄糖含量') 19 plt.ylabel('人数') 20 # 绘制葡萄糖——患病关系回归图 21 plt.subplot(2, 2, 4) 22 sns.regplot(x='gluc', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 23 plt.title('回归图 - 葡萄糖含量与患病关系') 24 plt.xlabel('葡萄糖含量') 25 plt.ylabel('患病情况') 26 # 调整子图之间的间距 27 plt.tight_layout() 28 plt.show()
结果:
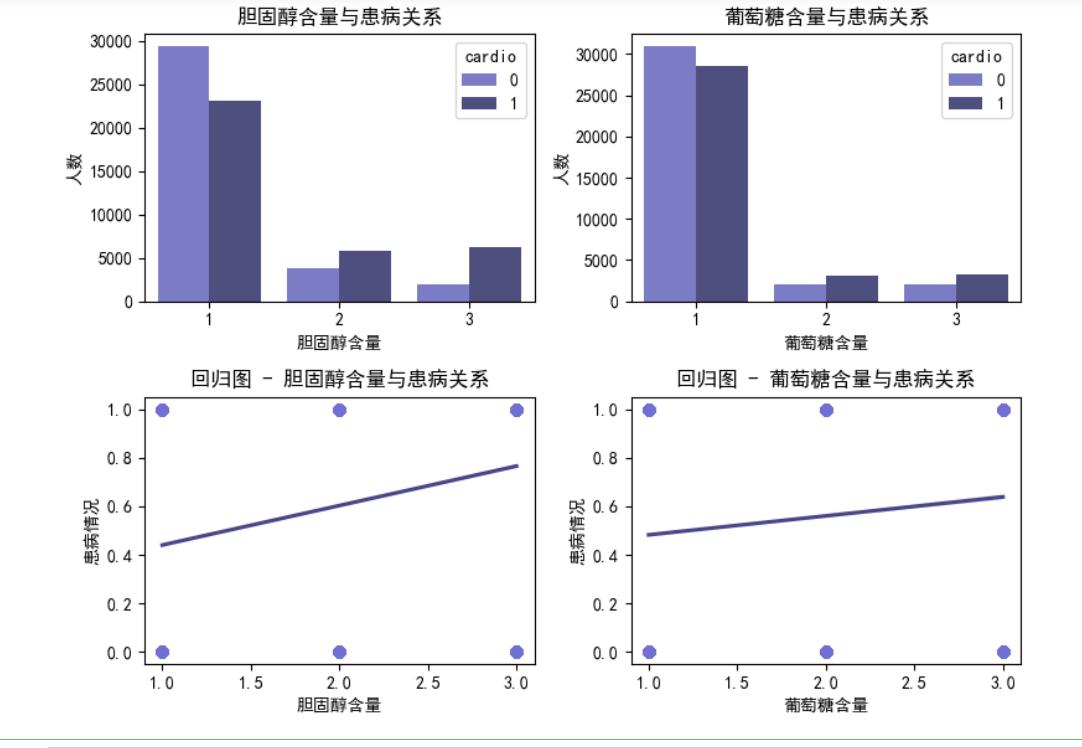
通过柱状图和回归图可以看出,胆固醇含量越高,患心血管疾病地几率越大,尤其是当胆固醇为3时,患病几率超过了50%。而葡萄糖含量与患病的几率的关系相对平衡,但同样也是葡萄糖含量越高,患心血管疾病的几率越高。胆固醇含量和葡萄糖含量是患心血管疾病的主要因素之一,而这两者的含量超标同样也可能导致患上糖尿病等疾病。
5.BMI值与患病关系
1 # 由于BMI数据中存在离群值,不是合理的BMI数值,因此要剔除 2 CVD_data = CVD_data[CVD_data['BMI'] <= 70] 3 4 plt.figure(figsize=(15, 12)) 5 # 直方图 6 plt.subplot(3, 2, 1) 7 plt.xticks(np.arange(0, 70, 5)) 8 sns.histplot(x='BMI', data=CVD_data[CVD_data['cardio'] == 1], kde=True) 9 plt.title('BMI值与患病情况关系 - 直方图') 10 plt.xlabel('BMI值') 11 plt.ylabel('频数') 12 # 核密度图 13 plt.subplot(3, 2, 2) 14 plt.xticks(np.arange(0, 70, 5)) 15 sns.kdeplot(x='BMI', data=CVD_data, hue='cardio', fill=True) 16 plt.title('BMI值与患病情况关系 - 核密度图') 17 plt.xlabel('BMI值') 18 plt.ylabel('密度') 19 # 回归图 20 plt.subplot(3, 2, 3) 21 plt.xticks(np.arange(0, 70, 5)) 22 sns.regplot(x='BMI', y='cardio', data=CVD_data, logistic=True) 23 plt.title('BMI值与患病情况关系 - 回归图') 24 plt.xlabel('BMI值') 25 plt.ylabel('患病情况') 26 # 盒图 27 plt.subplot(3, 2, 4) 28 sns.boxplot(x='cardio', y='BMI', data=CVD_data) 29 plt.title('BMI值与患病情况关系 - 盒图') 30 plt.xlabel('患病情况') 31 plt.ylabel('BMI值') 32 # 小提琴图 33 plt.subplot(3, 2, 5) 34 sns.violinplot(x='cardio', y='BMI', data=CVD_data, palette=['#ff793f', '#2c2c54']) 35 plt.title('BMI与患病关系的小提琴图') 36 plt.xlabel('患病情况') 37 plt.ylabel('BMI') 38 plt.tight_layout() 39 plt.show()
结果:

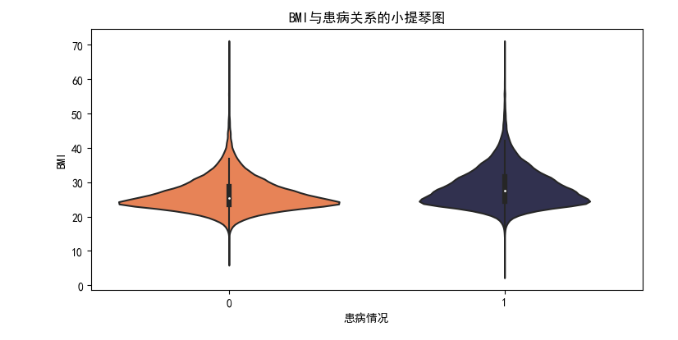
BMI不仅与患病几率有关系,同样和胆固醇,以及葡萄糖含量也有联系。从以上的图可以看出,BMI值为25时,患病人数到达峰值,随后开始下降。根据世界卫生组织的标准,BMI值大于24时即为超重,而28以上为肥胖(BMI值对健康地定义也会因人而异),因此可以看出,肥胖更容易导致患上心血管疾病,通过回归图也可以更加直观地看出这个趋势。除此之外,肥胖也更容易导致患上其他疾病。
6.收缩压与患病关系
有离群值,且数值不合理,因此需要剔除离群值
1 # 处理异常值,删除ap_hi列中数值大于300或小于1的行 2 CVD_data = CVD_data[(CVD_data['ap_hi'] <= 300) & (CVD_data['ap_hi'] >= 1)] 3 4 plt.figure(figsize=(10, 8)) 5 # 核密度图 6 plt.subplot(2, 2, 1) 7 plt.xticks(np.arange(0, 300, 25)) 8 sns.kdeplot(data=CVD_data, x='ap_hi', hue='cardio', fill=True, common_norm=False, palette=['#ff793f', '#2c2c54']) 9 plt.title('核密度图 - 收缩压与患病关系') 10 # 回归图 11 plt.subplot(2, 2, 2) 12 plt.xticks(np.arange(0, 300, 25)) 13 sns.regplot(x='ap_hi', y='cardio', data=CVD_data, scatter_kws={'color': '#2c2c54'}, line_kws={'color': '#ff793f'}) 14 plt.title('回归图 - 收缩压与患病关系') 15 # 箱图 16 plt.subplot(2, 2, 3) 17 plt.yticks(np.arange(0, 300, 25)) 18 sns.boxplot(x='cardio', y='ap_hi', data=CVD_data, palette=['#ff793f', '#2c2c54']) 19 plt.title('箱图 - 收缩压与患病关系') 20 # 小提琴图 21 plt.subplot(2, 2, 4) 22 plt.yticks(np.arange(0, 300, 25)) 23 sns.violinplot(x='cardio', y='ap_hi', data=CVD_data, palette=['#ff793f', '#2c2c54']) 24 plt.title('小提琴图 - 收缩压与患病关系') 25 plt.tight_layout() 26 plt.show()
结果:
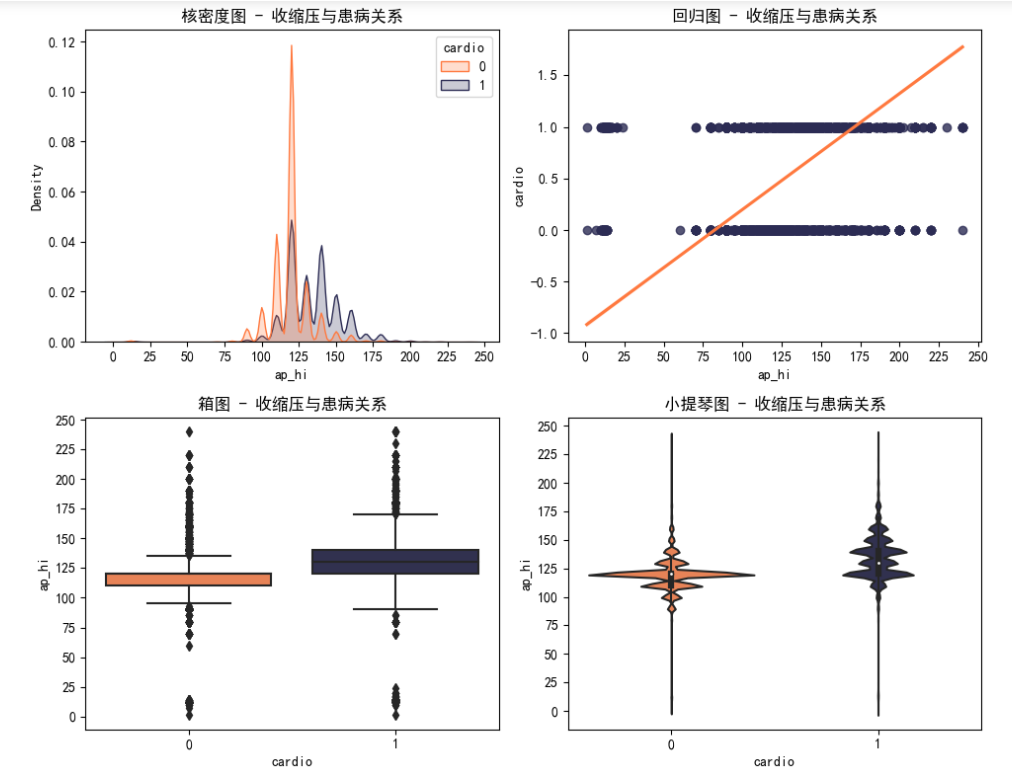
7.舒张压与患病关系
1 # 剔除离群值,删除ap_hi列中数值大于300或小于1的行 2 CVD_data = CVD_data[(CVD_data['ap_lo'] <= 300) & (CVD_data['ap_lo'] >= 1)] 3 4 plt.figure(figsize=(10, 8)) 5 # 核密度图 6 plt.subplot(2, 2, 1) 7 sns.kdeplot(data=CVD_data, x='ap_lo', hue='cardio', fill=True, common_norm=False, palette=['#ff793f', '#2c2c54']) 8 plt.title('核密度图 - 舒张压与患病关系') 9 # 回归图 10 plt.subplot(2, 2, 2) 11 sns.regplot(x='ap_lo', y='cardio', data=CVD_data, scatter_kws={'color': '#2c2c54'}, line_kws={'color': '#ff793f'}) 12 plt.title('回归图 - 舒张压与患病关系') 13 # 箱图 14 plt.subplot(2, 2, 3) 15 sns.boxplot(x='cardio', y='ap_lo', data=CVD_data, palette=['#ff793f', '#2c2c54']) 16 plt.title('箱图 - 舒张压与患病关系') 17 # 小提琴图 18 plt.subplot(2, 2, 4) 19 sns.violinplot(x='cardio', y='ap_lo', data=CVD_data, palette=['#ff793f', '#2c2c54']) 20 plt.title('小提琴图 - 舒张压与患病关系') 21 plt.tight_layout() 22 plt.show()
结果:
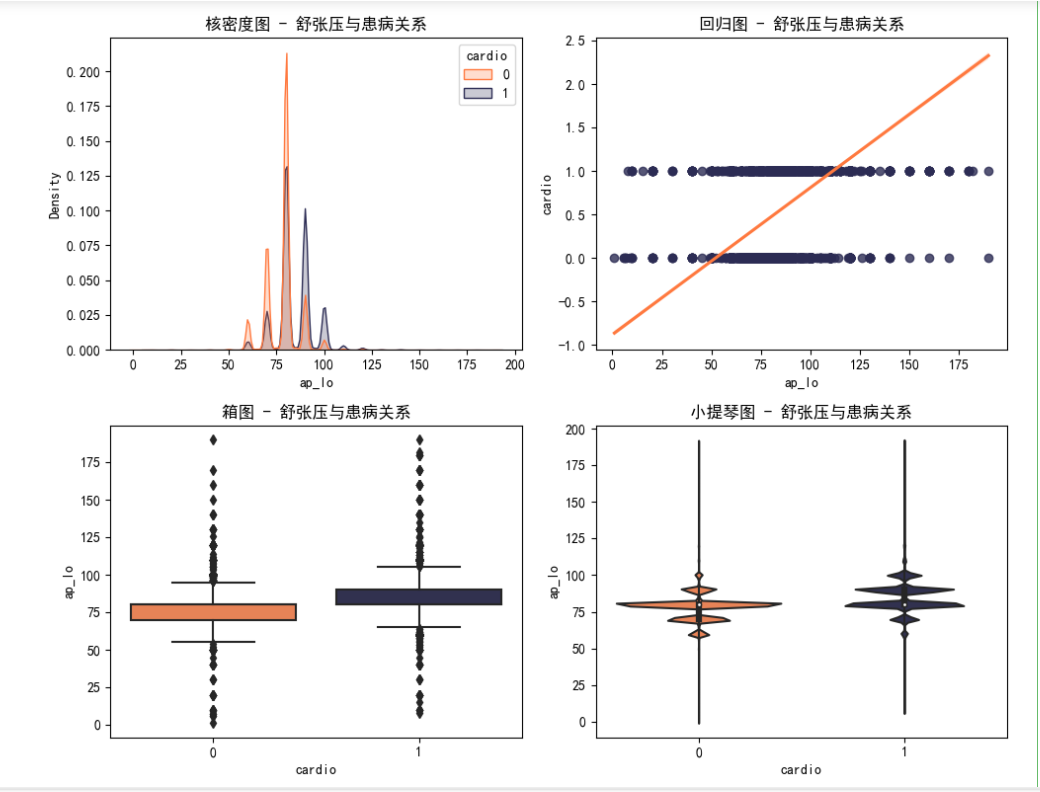
通过对收缩压和舒张压地数据图可以看出,高血压也是心血管疾病的主要诱因之一,并且相比其他因素尤为明显,血压越高,患心血管疾病的几率越大。因此,控制血压是预防心血管疾病最重要的手段。
8.(生活习惯)抽烟、酒精摄入、运动与患病情况的关系
1 plt.figure(figsize=(8, 6)) 2 # 绘制抽烟——患病关系柱状图 3 plt.subplot(2, 3, 1) 4 sns.countplot(x=CVD_data['smoke'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 5 plt.title('抽烟与患病关系') 6 plt.xlabel('是否抽烟') 7 plt.ylabel('人数') 8 # 绘制回归图 9 plt.subplot(2, 3, 4) 10 sns.regplot(x='smoke', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 11 plt.title('回归图 - 抽烟与患病关系') 12 plt.xlabel('抽烟情况') 13 plt.ylabel('患病情况') 14 # 绘制喝酒——患病关系柱状图 15 plt.subplot(2, 3, 2) 16 sns.countplot(x=CVD_data['alco'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 17 plt.title('喝酒与患病关系') 18 plt.xlabel('喝酒情况') 19 plt.ylabel('人数') 20 # 绘制回归图 21 plt.subplot(2, 3, 5) 22 sns.regplot(x='alco', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 23 plt.title('回归图 - 喝酒与患病关系') 24 plt.xlabel('喝酒情况') 25 plt.ylabel('患病情况') 26 # 绘制运动——患病关系柱状图 27 plt.subplot(2, 3, 3) 28 sns.countplot(x=CVD_data['active'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 29 plt.title('运动与患病关系') 30 plt.xlabel('运动情况') 31 plt.ylabel('人数') 32 # 绘制回归图 33 plt.subplot(2, 3, 6) 34 sns.regplot(x='active', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 35 plt.title('回归图 - 运动与患病关系') 36 plt.xlabel('运动情况') 37 plt.ylabel('患病情况') 38 # 调整子图之间的间距 39 plt.tight_layout() 40 plt.show()
结果:

通过柱状图可以看出,心血管疾病患者中,没有抽烟和酒精摄入的人数反而更多,所有抽烟和酒精摄入与患病几率没有多大关系,但根据研究和趋势,抽烟和究竟摄入也在增加心血管疾病的风险。而没有运动的人比有运动的人更容易患心血管疾病,因此,运动是预防心血管疾病的手段之一,通过回归图也能直观地看出趋势。
9.划分训练集和测试集
1 # 划分训练集和测试集 2 x_train, x_test, y_train, y_test = train_test_split(features, target, test_size=0.3, random_state=42)
3.预测模型
1.决策树
1 from sklearn.model_selection import train_test_split 2 from sklearn.tree import DecisionTreeClassifier 3 from sklearn.metrics import accuracy_score, classification_report, confusion_matrix 4 5 # 特征选择 6 features = CVD_data[['age', 'gender', 'height', 'weight', 'ap_hi', 'ap_lo', 'cholesterol', 'gluc', 'smoke', 'alco', 'active', 'BMI']] 7 # 目标变量 8 target = CVD_data['cardio'] 9 # 构建决策树模型 10 model = DecisionTreeClassifier(random_state=42) 11 model.fit(x_train, y_train) 12 # 在测试集上进行预测 13 y_pred = model.predict(x_test) 14 # 评估模型性能 15 accuracy = accuracy_score(y_test, y_pred) 16 conf_matrix = confusion_matrix(y_test, y_pred) 17 classification_rep = classification_report(y_test, y_pred) 18 # 打印模型性能指标 19 print(f'Accuracy: {accuracy:.2f}') 20 print(f'Classification Report:\n{classification_rep}')
模型精确度:
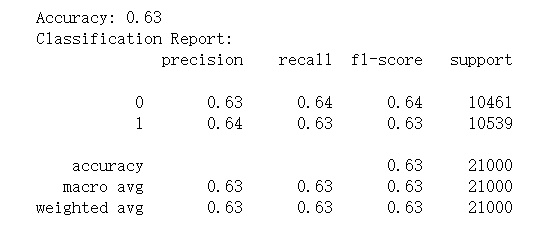
2.随机森林
1 from sklearn.ensemble import RandomForestClassifier 2 from sklearn.metrics import accuracy_score, classification_report, confusion_matrix 3 import matplotlib.pyplot as plt 4 from sklearn import tree 5 6 # 构建随机森林模型 7 model_rf = RandomForestClassifier(n_estimators=100, random_state=42) 8 model_rf.fit(x_train, y_train) 9 # 在测试集上进行预测 10 y_pred_rf = model_rf.predict(x_test) 11 # 评估模型性能 12 accuracy_rf = accuracy_score(y_test, y_pred_rf) 13 conf_matrix_rf = confusion_matrix(y_test, y_pred_rf) 14 classification_rep_rf = classification_report(y_test, y_pred_rf) 15 # 打印模型性能指标 16 print(f'Random Forest Accuracy: {accuracy_rf:.2f}') 17 print(f'Random Forest Classification Report:\n{classification_rep_rf}')
模型精确度:

4.完整代码
1 import pandas as pd 2 3 CVD_data = pd.read_csv('F:\JupyterNotebookFile\data\心血管疾病数据集.csv') 4 print(CVD_data.shape) # 输出数据集的维度 5 CVD_data.head() 6 # id列的数据序列存在间断不连续,无法作为索引,因此无实际作用,可删除 7 delete_column = 'id' 8 CVD_data.drop(delete_column, axis=1, inplace=True) 9 print(CVD_data.shape) 10 CVD_data.head() 11 # 数据集信息 12 CVD_data.info() 13 # 考虑到存在闰年和平年,取平均天数365.25天更精准,并且结果向下取整 14 import numpy as np 15 CVD_data['age'] = (np.floor(CVD_data['age'] / 365.25).astype(int)) 16 CVD_data['age'].head() 17 18 # 计算BMI值,添加"BMI"列 19 CVD_data["BMI"] = (CVD_data["weight"] / (CVD_data["height"] / 100)**2).round(1) 20 CVD_data.sample(3) 21 22 # 保存处理后的数据为新的CSV文件 23 CVD_data.to_csv('F:\JupyterNotebookFile\data\心血管疾病数据集(数据已处理).csv', index=False) 24 # 导入已处理完的数据集 25 CVD_data = pd.read_csv('F:\JupyterNotebookFile\data\心血管疾病数据集(数据已处理).csv') 26 print(CVD_data.shape) # 输出数据集的维度 27 CVD_data.head() 28 # 对数据的各个特征进行统计 29 data_summary = CVD_data.describe() 30 display(data_summary) 31 # 各个字段的数据分布情况 32 import matplotlib.pyplot as plt 33 CVD_data.hist(bins = 20, figsize = (40, 30)) 34 plt.show() 35 # 各字段的相关性 36 plt.figure(figsize = (12,10)) 37 sns.heatmap(CVD_data.corr(), annot =True) 38 plt.show() 39 40 # 患病、未患病,男性、女性占比分析 41 countHavaCVD = len(CVD_data[CVD_data.cardio == 1]) 42 countNoCVD = len(CVD_data[CVD_data.cardio == 0]) 43 countMale = len(CVD_data[CVD_data.gender == 1]) 44 countFemale = len(CVD_data[CVD_data.gender == 2]) 45 proportion_CVD = ("{:.2f}%".format((countHavaCVD / (len(CVD_data.cardio))*100))) 46 proportion_NoCVD = ("{:.2f}%".format((countNoCVD / (len(CVD_data.cardio))*100))) 47 proprotion_Male = ("{:.2f}%".format((countMale / (len(CVD_data.gender))*100))) 48 proportion_Female = ("{:.2f}%".format((countFemale / (len(CVD_data.gender))*100))) 49 50 result_data = pd.DataFrame({ 51 ' ': ['样本数量', '占比'], 52 '患病': [countHavaCVD,proportion_CVD], 53 '未患病': [countNoCVD, proportion_NoCVD], 54 '男性': [countMale, proprotion_Male], 55 '女性': [countFemale, proportion_Female] 56 }) 57 58 display(result_data) 59 60 import seaborn as sns 61 62 plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来正常显示中文标签 63 plt.rcParams['axes.unicode_minus'] = False # 用来正常显示负号 64 # 创建画布 65 fig = plt.figure(figsize = (8, 8)) 66 a = [countHavaCVD, countNoCVD] 67 b = [countMale, countFemale] 68 label1 = ['患病人数','未患病人数'] 69 label2 = ['男性','女性'] 70 #间隔 71 explode=(0,0.1) 72 #颜色 73 colors = ['#6a89cc','#1e3799'] 74 plt.subplot(1, 2, 1) 75 plt.pie(a,labels=label1, explode=explode ,colors=colors, autopct='%.2f%%',shadow=False,startangle=170) 76 plt.title('患病、未患病的人数比例') 77 #颜色 78 colors = ['#38ada9', '#78e08f'] 79 plt.subplot(1, 2, 2) 80 plt.pie(b,labels=label2,explode=explode ,colors=colors, autopct='%.2f%%',shadow=False,startangle=230) 81 plt.title('男性、女性的人数比例') 82 plt.show() 83 84 # 计算男性患者在男性总人数中的占比 85 ratioMaleCVD = len(CVD_data[(CVD_data.gender == 1) & (CVD_data.cardio == 1)]) / countMale 86 # 计算女性患者在女性总人数中的占比 87 ratioFemaleCVD = len(CVD_data[(CVD_data.gender == 2) & (CVD_data.cardio == 1)]) / countFemale 88 89 fig = plt.figure(figsize = (8, 8)) 90 labels_Male_CVD = ['男性患病', '男性未患病'] 91 sizes_Male_CVD = [ratioMaleCVD, 1 - ratioMaleCVD] 92 plt.subplot(1, 2, 1) 93 plt.pie(sizes_Male_CVD, labels=labels_Male_CVD, colors=['#f1c400', '#f39c12'], autopct='%1.2f%%', startangle=50, explode=explode) 94 plt.title('男性患病人数占比') 95 96 labels_Female_CVD = ['女性患病', '女性未患病'] 97 sizes_Female_CVD = [ratioFemaleCVD, 1 - ratioFemaleCVD] 98 plt.subplot(1, 2, 2) 99 plt.pie(sizes_Female_CVD, labels=labels_Female_CVD, colors=['#3F51B5', '#2196F3'], autopct='%1.2f%%', startangle=700, explode=explode) 100 plt.title('女性患病人数占比') 101 plt.show() 102 103 # 统计每个年龄的未患病和患病人数 104 counts_noCVD = CVD_data[CVD_data['cardio'] == 0].groupby('age').size() 105 counts_CVD = CVD_data[CVD_data['cardio'] == 1].groupby('age').size() 106 107 plt.figure(figsize=(12, 14)) 108 # 绘制折线图 109 plt.subplot(3, 2, 1) 110 plt.plot(counts_noCVD.index, counts_noCVD.values, label='未患病', color='#ff793f') 111 plt.plot(counts_CVD.index, counts_CVD.values, label='患病', color='#2c2c54') 112 plt.title('年龄与患病人数关系') 113 plt.xlabel('年龄') 114 plt.ylabel('患病人数') 115 plt.legend() 116 # 绘制直方图 117 plt.subplot(3, 2, 2) 118 sns.histplot(x=CVD_data['age'], kde=True, color='#2c2c54') 119 plt.title('年龄与患病关系的直方图') 120 plt.xlabel('年龄') 121 plt.ylabel('人数') 122 # 绘制小提琴图 123 plt.subplot(3, 2, 3) 124 sns.violinplot(x='cardio', y='age', data=CVD_data, palette=['#ff793f', '#2c2c54']) 125 plt.title('年龄与患病关系的小提琴图') 126 plt.xlabel('患病情况') 127 plt.ylabel('年龄') 128 plt.subplot(3, 2, 4) 129 # 绘制核密度图 130 sns.kdeplot(x='age', data=CVD_data[CVD_data['cardio'] == 0], color='#ff793f', label='未患病', fill=True) 131 sns.kdeplot(x='age', data=CVD_data[CVD_data['cardio'] == 1], color='#2c2c54', label='患病', fill=True) 132 plt.title('年龄与患病关系的核密度图') 133 plt.xlabel('年龄') 134 plt.ylabel('核密度估计') 135 # 绘制盒图 136 plt.subplot(3, 2, 5) 137 sns.boxplot(x='cardio', y='age', data=CVD_data, palette=['#ff793f', '#2c2c54']) 138 plt.title('年龄与患病关系的盒图') 139 plt.xlabel('患病情况') 140 plt.ylabel('年龄') 141 # 绘制散点图 142 plt.subplot(3, 2, 6) 143 sns.regplot(x='age',y='cardio',data=CVD_data, color='#2c2c54') 144 plt.title('年龄与患病关系的散点图') 145 plt.xlabel('年龄') 146 plt.ylabel('患病情况') 147 plt.tight_layout() 148 plt.show() 149 150 plt.figure(figsize=(8, 6)) 151 # 绘制胆固醇——患病关系柱状图 152 plt.subplot(2, 2, 1) 153 sns.countplot(x=CVD_data['cholesterol'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 154 plt.title('胆固醇含量与患病关系') 155 plt.xlabel('胆固醇含量') 156 plt.ylabel('人数') 157 # 绘制胆固醇——患病关系回归图 158 plt.subplot(2, 2, 3) 159 sns.regplot(x='cholesterol', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 160 plt.title('回归图 - 胆固醇含量与患病关系') 161 plt.xlabel('胆固醇含量') 162 plt.ylabel('患病情况') 163 # 绘制葡萄糖——患病关系柱状图 164 plt.subplot(2, 2, 2) 165 sns.countplot(x=CVD_data['gluc'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 166 plt.title('葡萄糖含量与患病关系') 167 plt.xlabel('葡萄糖含量') 168 plt.ylabel('人数') 169 # 绘制葡萄糖——患病关系回归图 170 plt.subplot(2, 2, 4) 171 sns.regplot(x='gluc', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 172 plt.title('回归图 - 葡萄糖含量与患病关系') 173 plt.xlabel('葡萄糖含量') 174 plt.ylabel('患病情况') 175 # 调整子图之间的间距 176 plt.tight_layout() 177 plt.show() 178 179 # 由于BMI数据中存在离群值,不是合理的BMI数值,因此要剔除 180 CVD_data = CVD_data[CVD_data['BMI'] <= 70] 181 182 plt.figure(figsize=(15, 12)) 183 # 直方图 184 plt.subplot(3, 2, 1) 185 plt.xticks(np.arange(0, 70, 5)) 186 sns.histplot(x='BMI', data=CVD_data[CVD_data['cardio'] == 1], kde=True) 187 plt.title('BMI值与患病情况关系 - 直方图') 188 plt.xlabel('BMI值') 189 plt.ylabel('频数') 190 # 核密度图 191 plt.subplot(3, 2, 2) 192 plt.xticks(np.arange(0, 70, 5)) 193 sns.kdeplot(x='BMI', data=CVD_data, hue='cardio', fill=True) 194 plt.title('BMI值与患病情况关系 - 核密度图') 195 plt.xlabel('BMI值') 196 plt.ylabel('密度') 197 # 回归图 198 plt.subplot(3, 2, 3) 199 plt.xticks(np.arange(0, 70, 5)) 200 sns.regplot(x='BMI', y='cardio', data=CVD_data, logistic=True) 201 plt.title('BMI值与患病情况关系 - 回归图') 202 plt.xlabel('BMI值') 203 plt.ylabel('患病情况') 204 # 盒图 205 plt.subplot(3, 2, 4) 206 sns.boxplot(x='cardio', y='BMI', data=CVD_data) 207 plt.title('BMI值与患病情况关系 - 盒图') 208 plt.xlabel('患病情况') 209 plt.ylabel('BMI值') 210 # 小提琴图 211 plt.subplot(3, 2, 5) 212 sns.violinplot(x='cardio', y='BMI', data=CVD_data, palette=['#ff793f', '#2c2c54']) 213 plt.title('BMI与患病关系的小提琴图') 214 plt.xlabel('患病情况') 215 plt.ylabel('BMI') 216 plt.tight_layout() 217 plt.show() 218 219 # 处理异常值,删除ap_hi列中数值大于300或小于1的行 220 CVD_data = CVD_data[(CVD_data['ap_hi'] <= 300) & (CVD_data['ap_hi'] >= 1)] 221 222 plt.figure(figsize=(10, 8)) 223 # 核密度图 224 plt.subplot(2, 2, 1) 225 plt.xticks(np.arange(0, 300, 25)) 226 sns.kdeplot(data=CVD_data, x='ap_hi', hue='cardio', fill=True, common_norm=False, palette=['#ff793f', '#2c2c54']) 227 plt.title('核密度图 - 收缩压与患病关系') 228 # 回归图 229 plt.subplot(2, 2, 2) 230 plt.xticks(np.arange(0, 300, 25)) 231 sns.regplot(x='ap_hi', y='cardio', data=CVD_data, scatter_kws={'color': '#2c2c54'}, line_kws={'color': '#ff793f'}) 232 plt.title('回归图 - 收缩压与患病关系') 233 # 箱图 234 plt.subplot(2, 2, 3) 235 plt.yticks(np.arange(0, 300, 25)) 236 sns.boxplot(x='cardio', y='ap_hi', data=CVD_data, palette=['#ff793f', '#2c2c54']) 237 plt.title('箱图 - 收缩压与患病关系') 238 # 小提琴图 239 plt.subplot(2, 2, 4) 240 plt.yticks(np.arange(0, 300, 25)) 241 sns.violinplot(x='cardio', y='ap_hi', data=CVD_data, palette=['#ff793f', '#2c2c54']) 242 plt.title('小提琴图 - 收缩压与患病关系') 243 plt.tight_layout() 244 plt.show() 245 246 # 剔除离群值,删除ap_hi列中数值大于300或小于1的行 247 CVD_data = CVD_data[(CVD_data['ap_lo'] <= 300) & (CVD_data['ap_lo'] >= 1)] 248 249 plt.figure(figsize=(10, 8)) 250 # 核密度图 251 plt.subplot(2, 2, 1) 252 sns.kdeplot(data=CVD_data, x='ap_lo', hue='cardio', fill=True, common_norm=False, palette=['#ff793f', '#2c2c54']) 253 plt.title('核密度图 - 舒张压与患病关系') 254 # 回归图 255 plt.subplot(2, 2, 2) 256 sns.regplot(x='ap_lo', y='cardio', data=CVD_data, scatter_kws={'color': '#2c2c54'}, line_kws={'color': '#ff793f'}) 257 plt.title('回归图 - 舒张压与患病关系') 258 # 箱图 259 plt.subplot(2, 2, 3) 260 sns.boxplot(x='cardio', y='ap_lo', data=CVD_data, palette=['#ff793f', '#2c2c54']) 261 plt.title('箱图 - 舒张压与患病关系') 262 # 小提琴图 263 plt.subplot(2, 2, 4) 264 sns.violinplot(x='cardio', y='ap_lo', data=CVD_data, palette=['#ff793f', '#2c2c54']) 265 plt.title('小提琴图 - 舒张压与患病关系') 266 plt.tight_layout() 267 plt.show() 268 269 plt.figure(figsize=(8, 6)) 270 # 绘制抽烟——患病关系柱状图 271 plt.subplot(2, 3, 1) 272 sns.countplot(x=CVD_data['smoke'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 273 plt.title('抽烟与患病关系') 274 plt.xlabel('是否抽烟') 275 plt.ylabel('人数') 276 # 绘制回归图 277 plt.subplot(2, 3, 4) 278 sns.regplot(x='smoke', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 279 plt.title('回归图 - 抽烟与患病关系') 280 plt.xlabel('抽烟情况') 281 plt.ylabel('患病情况') 282 # 绘制喝酒——患病关系柱状图 283 plt.subplot(2, 3, 2) 284 sns.countplot(x=CVD_data['alco'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 285 plt.title('喝酒与患病关系') 286 plt.xlabel('喝酒情况') 287 plt.ylabel('人数') 288 # 绘制回归图 289 plt.subplot(2, 3, 5) 290 sns.regplot(x='alco', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 291 plt.title('回归图 - 喝酒与患病关系') 292 plt.xlabel('喝酒情况') 293 plt.ylabel('患病情况') 294 # 绘制运动——患病关系柱状图 295 plt.subplot(2, 3, 3) 296 sns.countplot(x=CVD_data['active'], hue=CVD_data['cardio'], palette=['#706fd3', '#474787']) 297 plt.title('运动与患病关系') 298 plt.xlabel('运动情况') 299 plt.ylabel('人数') 300 # 绘制回归图 301 plt.subplot(2, 3, 6) 302 sns.regplot(x='active', y='cardio', data=CVD_data, scatter_kws={'color': '#706fd3'}, line_kws={'color': '#474787'}) 303 plt.title('回归图 - 运动与患病关系') 304 plt.xlabel('运动情况') 305 plt.ylabel('患病情况') 306 # 调整子图之间的间距 307 plt.tight_layout() 308 plt.show() 309 310 # 将划分训练集和测试集 311 x_train, x_test, y_train, y_test = train_test_split(features, target, test_size=0.3, random_state=42) 312 313 # 决策树 314 from sklearn.model_selection import train_test_split 315 from sklearn.tree import DecisionTreeClassifier 316 from sklearn.metrics import accuracy_score, classification_report, confusion_matrix 317 318 # 特征选择 319 features = CVD_data[['age', 'gender', 'height', 'weight', 'ap_hi', 'ap_lo', 'cholesterol', 'gluc', 'smoke', 'alco', 'active', 'BMI']] 320 # 目标变量 321 target = CVD_data['cardio'] 322 # 构建决策树模型 323 model = DecisionTreeClassifier(random_state=42) 324 model.fit(x_train, y_train) 325 # 在测试集上进行预测 326 y_pred = model.predict(x_test) 327 # 评估模型性能 328 accuracy = accuracy_score(y_test, y_pred) 329 conf_matrix = confusion_matrix(y_test, y_pred) 330 classification_rep = classification_report(y_test, y_pred) 331 # 打印模型性能指标 332 print(f'Accuracy: {accuracy:.2f}') 333 print(f'Classification Report:\n{classification_rep}') 334 335 # 随机森林 336 from sklearn.ensemble import RandomForestClassifier 337 from sklearn.metrics import accuracy_score, classification_report, confusion_matrix 338 import matplotlib.pyplot as plt 339 from sklearn import tree 340 341 # 构建随机森林模型 342 model_rf = RandomForestClassifier(n_estimators=100, random_state=42) 343 model_rf.fit(x_train, y_train) 344 # 在测试集上进行预测 345 y_pred_rf = model_rf.predict(x_test) 346 # 评估模型性能 347 accuracy_rf = accuracy_score(y_test, y_pred_rf) 348 conf_matrix_rf = confusion_matrix(y_test, y_pred_rf) 349 classification_rep_rf = classification_report(y_test, y_pred_rf) 350 # 打印模型性能指标 351 print(f'Random Forest Accuracy: {accuracy_rf:.2f}') 352 print(f'Random Forest Classification Report:\n{classification_rep_rf}')
四、总结
通过数据挖掘,对各个指标与患心血管疾病几率的相关性的分析,总结出了几个导致心血管疾病的重要因素,包括年龄大、高血压、肥胖,前两者基本上都会伴随出现,而如今,越来越多的年轻人也面临着高血压的危险,肥胖也逐渐在低龄化。此外,如今的人们因为工作的繁忙,或是沉迷手机,经常久坐,且很少运动,也会增加心血管疾病的风险。因此,在生活中应该保持健康的生活习惯,以及增加运动来预防心血管疾病。
在完成本次课程设计的过程中,我对大数据分析的整个过程有了更深入的了解,运用上课所学到的分析方法和实现技术,完成了大数据分析的流程,包括数据清洗、挖掘、数据可视化、分析,这一套完整的流程,环环相扣,在这过程中,我又学到了一些新的分析方法和思路。